Programme de recherche : Pathologies neuromusculaires et physiopathologie
Accueil > Équipe 5 > Programme de recherche > Axe 2
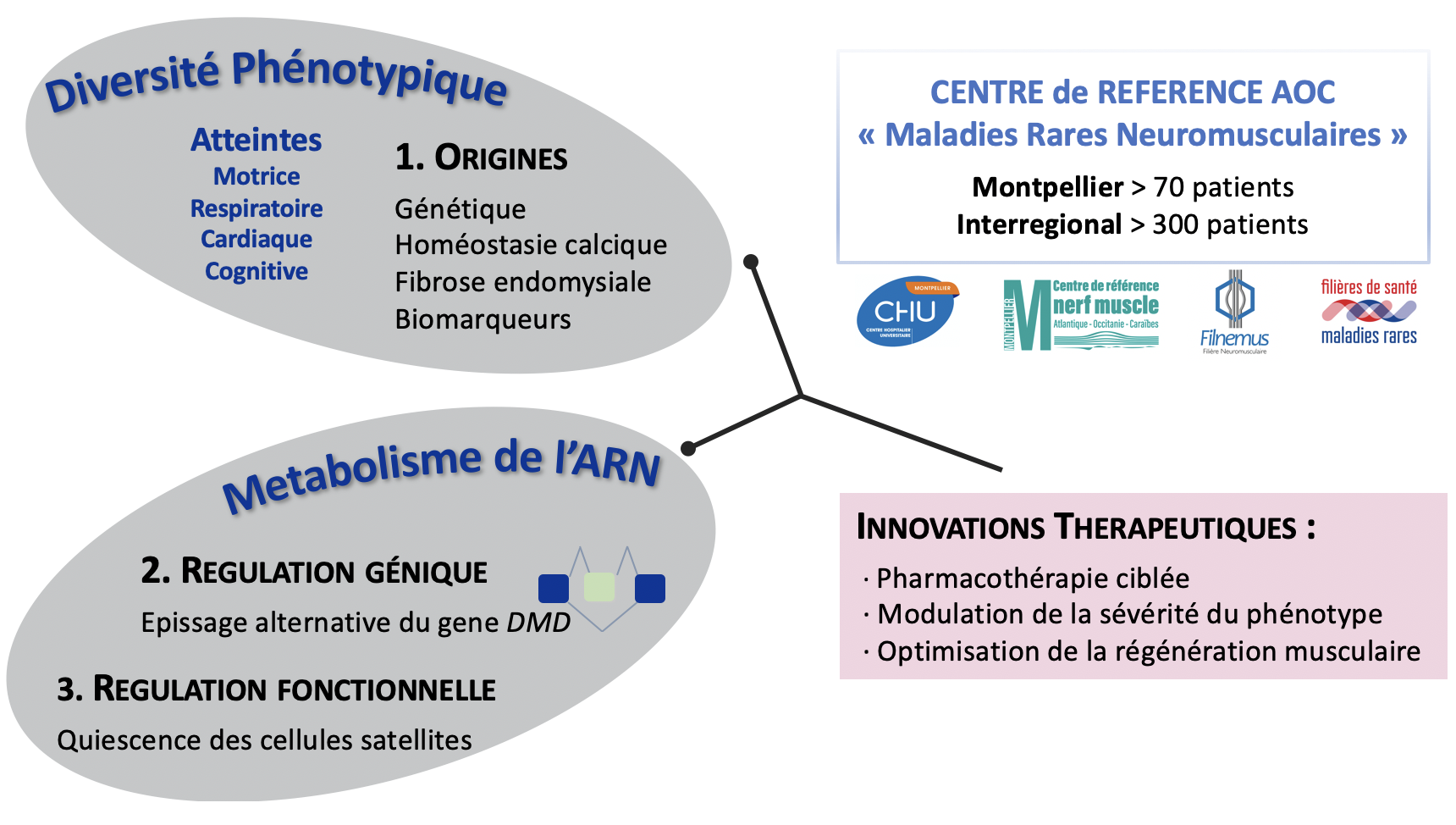
Dystrophinopathies : diversité phénotypique et régulation génique
C. Notarnicola, S. Tuffery-Giraud, J. Miro, P. Meyer, M. Cossée, F. Rivier
Les dystrophinopathies regroupent des formes cliniques différentes de dystrophie musculaire dues à des mutations dans le gène DMD incluant la dystrophie musculaire de Duchenne (DMD, forme sévère) et la dystrophie musculaire de Becker (BMD, forme modérée).
L’objectif de notre projet de recherche est d’étudier dans la cellule musculaire normale et/ou pathologique les mécanismes moléculaires et cellulaires impliqués dans (1) l’hétérogénéité clinique observée chez les patients, (2) la régulation de l’expression du gène DMD, et (3) la fonction et l’activation des cellules souches musculaires. Ces approches peuvent à terme contribuer à l’identification de biomarqueurs et au développement de thérapies ciblées.
Origine de la diversité phénotypique
L’histoire clinique de la DMD, bien qu’apparemment uniforme, cache une grande variabilité phénotypique, indépendante du type de mutations dans le gène DMD qui conduisent toutes à une absence de dystrophine dans le muscle squelettique (Humbertclaude et al. 2012). Une étude multicentrique INSERM-DGOS (étude FEDmd) est menée afin d’explorer différents mécanismes (homéostasie calcique, fibrose endomysiale musculaire, processus de régénération musculaire) pouvant contribuer à la variabilité clinique observée. Notre projet vise également à identifier des biomarqueurs diagnostiques et/ou pronostiques pertinents pour le suivi clinique ou l’évaluation de la réponse aux traitements dans la DMD, notamment des myomiR circulants grâce à une étude longitudinale réalisée dans une cohorte de patients (projet BioDystromiRs).
Mécanismes moléculaires de l’épissage du gène DMD
L’épissage alternatif des ARN pre-messagers est un processus essentiel de l’expression des gènes, qui repose sur la reconnaissance coordonnée de multiples signaux recrutant des protéines de liaison à l’ARN (RNA Binding Proteins, RBPs) ayant une action activatrice ou inhibitrice de l’épissage selon les besoins cellulaires. Notre projet vise à étudier la régulation de l’épissage du gène DMD (2.3 Mb, 79 exons, 7 promoteurs tissu-spécifique):
- En contexte physiologique : nous avons démontré par une approche de RNA-seq ciblé l’absence d’épissage alternatif dans le transcrit exprimé dans muscle squelettique adulte contrairement à ce qui est observé dans les isoformes cérébrales de DMD (Bougé et al. 2017). Un crible fonctionnel par RNAi nous a permis d’identifier des RBPs régulant l’inclusion d’exons DMD dans le transcrit musculaire (Miro et al. 2020). Notre objectif est maintenant d’explorer les mécanismes moléculaires impliqués grâce à une approche intégrant des analyses bioinformatiques, fonctionnelles et biochimiques afin de contribuer à meilleure connaissance de la régulation spatio-temporelle de l’expression du gène DMD.
- En contexte pathologique, nous nous intéressons au rôle de l’épissage alternatif comme modificateur de la sévérité du phénotype chez les patients DMD (Tuffery-Giraud et al. 2017). Nous étudions en particulier les sauts d’exon induits par des mutations non sens conduisant à une ré-expression partielle de dystrophine et une atténuation du phénotype chez les patients (Miro et al. 2015). Notre objectif est d’identifier des RBPs (notamment de la famille des SR protéines) pouvant être ciblées dans des approches pharmacologiques dans le but de potentialiser le saut d’exon spontané observé chez le patient et améliorer la correction du phénotype.
Régulation de la fonction des cellules satellites
Le muscle squelettique a une capacité remarquable de régénération après une lésion sévère grâce à la présence, sous la lame basale des myofibrilles, des cellules souches musculaires appelées cellules satellites (SC). Certaines pathologies dégénératives comme la DMD affectent la fonction des SC et par conséquent altère la régénération musculaire. Notre projet de recherche vise à étudier les mécanismes intrinsèques qui régulent la fonction des cellules souches musculaires adultes :
- En condition physiologique, nous nous intéressons plus particulièrement à la quiescence des SC, un stade requis pour la survie à long terme du pool de cellules souches et l’identité du tissu musculaire. En effet dans le muscle adulte, les SC sont quiescentes mais sont aussi capable d’activer très rapidement le cycle cellulaire et le programme myogénique, une fonction qui requiert une régulation spécifique des ARN messagers. Notre objectif est d’identifier les mécanismes moléculaires qui contrôlent la traduction des ARNm (RBP) pour maintenir la quiescence des SC mais aussi les voies de signalisation impliquées dans la synthèse protéique globale.
- En contexte pathologique : Pour assurer leurs fonctions, les SC doivent maintenir une balance dynamique entre les différents états cellulaires. Nous avons montré le rôle de l’acide rétinoïque et des voies de signalisation associées sur la régulation de l’immaturité des cellules musculaires précurseurs (El Haddad M. et al. 2017). Par analyses transcriptomiques sur les différents stades myogéniques, nous avons défini des profils d’expression géniques issus de patients DMD. Notre objectif est maintenant d’étudier la fonction des gènes qui régulent le devenir des SC dans un muscle dystrophique afin de développer des approches thérapeutiques pour préserver la fonction des SC et par conséquent la régénération musculaire.
Financements